Jeanne Ropars
Poste : Chargée de Recherche
Poste détaillé : Chargée de Recherche CNRS
Équipe : Génétique et Écologie Évolutives
Coordonnées :
Laboratoire Écologie, Société et Évolution – IDEEV
Bureau 2316 Bât. 680 – 12, route 128
91190 Gif Sur Yvette
Tél : +33 (0)1 69 15 46 93
Fax : –
Email : jeanne.ropars (at) universite-paris-saclay.fr
Est-ce que l'adaptation est répétable ?
Activités de recherche
Je cherche à comprendre les mécanismes de l’évolution et de l’adaptation, incluant les transferts horizontaux de gènes, la reproduction sexuée, les flux de gènes et la diversité génétique. Pour ce faire, j’utilise les champignons comme modèles car je pense qu’ils sont de bons modèles pour tester des hypothèses de biologie évolutive et développer de nouveau concepts. En effet, certains champignons possèdent de nombreux avantages expérimentaux : ils ont des petits génomes, ils sont facilement cultivables en conditions de laboratoire, ils ont des temps de génération courts, ils peuvent rester en vie longtemps congelés, ils sont faciles à transformer génétiquement et ils montrent une grande diversité en termes de lignées et de niches écologiques.
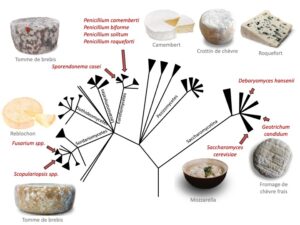
Ces caractéristiques permettent de répondre à des questions de biologie évolutive en utilisant des approches complémentaires, incluant les méthodes génomiques et expérimentales. En particulier, l’existence de multiples lignées de champignons distantes phylogénétiquement et utilisées dans la production du même type de nourriture procure un système modèle idéal pour étudier la répétabilité de l’évolution en fournissant plusieurs réplicas indépendants d’adaptation récente à une même niche écologique, sous sélection forte et récente. Depuis 2018, j’étudie la répétabilité de l’évolution en utilisant les champignons du fromage comme modèles, en me basant sur des analyses génomiques, historiques et des expériences.
Image : Mon projet de recherche : est-ce que l’adaptation est répétable ? Les champignons du fromage sont des modèles de choix pour étudier l’adaptation parallèle car 1) plusieurs lignées phylogénétiquement éloignées sont retrouvées dans la même niche écologique et 2) cette adaptation est le résultat d’une sélection forte et récente (< 8000 ans). Mes questions de recherche sont : Est-ce que ces adaptations parallèles ont menées à des phénotypes convergents ? Est-ce que les mêmes gènes/régions génomiques et les mêmes mécanismes génomiques sont impliqués dans ces adaptations parallèles ? Arbre modifié depuis Lifemap (http://lifemap.univ-lyon1.fr/).
Parcours
2022 : Habilitation à Diriger des Recherches, Université Paris-Saclay. Soutenue le 30 juin 2022 à Gif-sur-Yvette
2018- : Chargée de recherche CNRS, Université Paris-Saclay, Gif-sur-Yvette
2016-2018 : Post-Doc, Institut Pasteur, Paris, France – Laboratoire de C. d’Enfert, champignon pathogène humain Candida albicans
2014-2015 :Post-Doc, Université d’Ottawa, Ottawa, Canada – Laboratoire de N. Corradi, champignons arbusculaires mycorrhiziens
2012-2014 : Post-Doc, Université Paris-Sud XI, Orsay, France – Equipe de T. Giraud, champignon Penicillium du fromage
2009-2012 : Doctorat en biologie évolutive, Muséum National d’Histoire Naturelle, Paris, France – Directrice de thèse : J. Dupont. « Exploration de génomes d’inoculums fongiques utilisés en fromagerie, pour leur caractérisation systématique, la recherche de mécanismes adaptatifs et l’évaluation de la capacité à la reproduction sexuée chez Penicillium roqueforti ». Soutenue le 22 juin 2012 à Paris
2006-2008 : Master Systématique et Evolution, Muséum National d’Histoire Naturelle, Paris, France
Publications
Lo Y, Bruxaux J, Rodríguez de la Vega RC, O’Donnell S, Snirc A, Coton M, et al. Domestication in dry‐cured meat Penicillium fungi: Convergent specific phenotypes and horizontal gene transfers without strong genetic subdivision. Evol Appl. 2023;16:1637–60. https://onlinelibrary.wiley.com/doi/full/10.1111/eva.13591
Giraud T, Ropars J, Stukenbrock EH, Amato KR, Rodriguez de la Vega R. Evolutionary ecology of human-associated microbes. Mol Ecol. 2023;32(10):2369-2373. https://onlinelibrary.wiley.com/doi/abs/10.1111/mec.16966
Crequer E, Ropars J, Jany J-L, Caron T, Coton M, Snirc A, et al. A new cheese population in Penicillium roqueforti and adaptation of the five populations to their ecological niche. Evol Appl. 2023;00:1–20. https://onlinelibrary.wiley.com/doi/full/10.1111/eva.13578
Bennetot B, Vernadet J-P, Perkins V, Hautefeuille S, Rodríguez De La Vega RC, O’Donnell S, et al. Domestication of different varieties in the cheese-making fungus Geotrichum candidum. Peer Community J. 2023;3:e45. https://peercommunityjournal.org/articles/10.24072/pcjournal.266/
Giraud T, Ropars J. The variety of bread-making practices promotes diversity conservation in food microbial communities. Peer Community Evol Biol. 2022;100154. https://evolbiol.peercommunityin.org/articles/rec?id=533
Ropars J. Penicillium camemberti. Encycl Dairy Sci. Elsevier; 2022. p. 593–8.
Ropars J, Giraud T. Convergence in domesticated fungi used for cheese and dry-cured meat maturation: beneficial traits, genomic mechanisms, and degeneration. Curr Opin Microbiol. 2022;70:102236.
Savary O, Coton M, Frisvad J, Nodet P, Ropars J, Coton E, et al. Unexpected Nectriaceae species diversity in cheese, description of Bisifusarium allantoides sp. nov., Bisifusarium penicilloides sp. nov., Longinectria gen. nov. lagenoides sp. nov. and Longinextria verticilliforme sp. nov. Mycosphere. 2021;12:1077–100. https://www.mycosphere.org/pdf/MYCOSPHERE_12_1_13-1.pdf
Ropars J, Didiot E, Rodríguez de la Vega RC, Bennetot B, Coton M, Poirier E, et al. Domestication of the emblematic white cheese-making fungus Penicillium camemberti and its diversification into two varieties. Current Biology. 2020;30:4441–53. https://www.cell.com/current-biology/fulltext/S0960-9822(20)31272-0.
Ropars J, Caron T, Lo Y, Bennetot B, Giraud T. The domestication of Penicillium cheese fungi. Comptes Rendus Biologies. 2020;343:155–76. https://comptes-rendus.academie-sciences.fr/biologies/article/CRBIOL_2020__343_2_155_0.pdf
Hernández-Cervantes A, Znaidi S, van Wijlick L, Denega I, Basso V, Ropars J, et al. A conserved regulator controls asexual sporulation in the fungal pathogen Candida albicans. Nature Communications. 2020;11:6224. https://www.nature.com/articles/s41467-020-20010-9
Dumas E, Feurtey A, Vega RCR de la, Prieur SL, Snirc A, Coton M, et al. Independent domestication events in the blue-cheese fungus Penicillium roqueforti. Molecular Ecology. 2020;29:2639–60. https://onlinelibrary.wiley.com/doi/full/10.1111/mec.15359
Chen ECH, Mathieu S, Hoffrichter A, Ropars J, Dreissig S, Fuchs J, et al. More Filtering on SNP Calling Does Not Remove Evidence of Inter-Nucleus Recombination in Dikaryotic Arbuscular Mycorrhizal Fungi. Front Plant Sci 1.
Ropars J, Maufrais C, Diogo D, Marcet-Houben M, Perin A, Sertour N, et al. Gene flow contributes to diversification of the major fungal pathogen Candida albicans. Nature Communications. 2018;9:2253. https://www.nature.com/articles/s41467-018-04787-4
Chen EC, Mathieu S, Hoffrichter A, Sedzielewska-Toro K, Peart M, Pelin A, et al. Single nucleus sequencing reveals evidence of inter-nucleus recombination in arbuscular mycorrhizal fungi. Wittkopp PJ, Martin F, Geurts R, Young P, Seidl M, editors. eLife. 2018;7:e39813.
Chen ECH, Morin E, Beaudet D, Noel J, Yildirir G, Ndikumana S, et al. High intraspecific genome diversity in the model arbuscular mycorrhizal symbiont Rhizophagus irregularis. New Phytologist. 2018;220:1161–71.
Ropars J, López-Villavicencio M, Snirc A, Lacoste S, Giraud T. Blue cheese-making has shaped the population genetic structure of the mould Penicillium roqueforti. PLOS ONE. 2017;12:e0171387.
Dupont J, Dequin S, Giraud T, Le Tacon F, Marsit S, Ropars J, et al. Fungi as a Source of Food. Microbiology Spectrum. 2017; 5
d’Enfert C, Bougnoux M-E, Feri A, Legrand M, Loll-Krippleber R, Marton T, et al. Genome Diversity and Dynamics in Candida albicans. 2017; Pp. 205–232 in R. Prasad, ed. Candida albicans: Cellular and Molecular Biology. Springer International Publishing, Cham.
Ropars J, Toro KS, Noel J, Pelin A, Charron P, Farinelli L, et al. Evidence for the sexual origin of heterokaryosis in arbuscular mycorrhizal fungi. Nature Microbiology. 2016;16033.
Ropars J, Lo Y, Dumas E, Snirc A, Begerow D, Rollnik T, et al. Fertility depression among cheese‐making Penicillium roqueforti strains suggests degeneration during domestication. Evolution. 2016;70:2099–109.
Ropars J, de la Vega RCR, López-Villavicencio M, Gouzy J, Dupont J, Swennen D, et al. Diversity and Mechanisms of Genomic Adaptation in Penicillium. Aspergillus and Penicillium in the Post-genomic Era. 2016; P. 210 in Aspergillus and Penicillium in the Post-genomic Era. Ronald P. de Vries, Isabelle Benoit Gelber, Mikael Rordam Andersen.
Ropars J, Rodríguez de la Vega RC, López-Villavicencio M, Gouzy J, Sallet E, Dumas É, et al. Adaptive horizontal gene transfers between multiple cheese-associated Fungi. Current Biology. 2015;25:2562–9. https://www.cell.com/current-biology/pdfExtended/S0960-9822(15)00996-3
Ropars J, Corradi N. Homokaryotic vs heterokaryotic mycelium in arbuscular mycorrhizal fungi: different techniques, different results? New Phytologist. 2015;208:638–41.
Pintye A, Ropars J, Harvey N, Shin H-D, Leyronas C, Nicot PC, et al. Host Phenology and Geography as Drivers of Differentiation in Generalist Fungal Mycoparasites. Zhang Z, editor. PLOS ONE. 2015;10:e0120703.
Gillot G, Jany J-L, Coton M, Le Floch G, Debaets S, Ropars J, et al. Insights into Penicillium roqueforti morphological and genetic diversity. Lumbsch HT, editor. PLOS ONE. 2015;10:e0129849.
Cornille A, Feurtey A, Gélin U, Ropars J, Misvanderbrugge K, Gladieux P, et al. Anthropogenic and natural drivers of gene flow in a temperate wild fruit tree: a basis for conservation and breeding programs in apples. Evolutionary Applications. 2015;8:373–84.
Ropars J, López-Villavicencio M, Dupont J, Snirc A, Gillot G, Coton M, et al. Induction of sexual reproduction and genetic diversity in the cheese fungus Penicillium roqueforti. Evolutionary Applications. 2014;7:433–41.
Ropars J, Aguileta G, de Vienne DM, Giraud T. Massive gene swamping among cheese-making Penicillium fungi. Microbial Cell. 2014;1.
Gladieux P, Ropars J, Badouin H, Branca A, Aguileta G, de Vienne DM, et al. Fungal evolutionary genomics provides insight into the mechanisms of adaptive divergence in eukaryotes. Molecular Ecology. 2014;23:753–73.
Cheeseman K, Ropars J, Renault P, Dupont J, Gouzy J, Branca A, et al. Multiple recent horizontal transfers of a large genomic region in cheese making fungi. Nat Commun. 2014;5:2876.
Ropars J, Dupont J, Fontanillas E, Rodríguez de la Vega RC, Malagnac F, Coton M, et al. Sex in cheese: evidence for sexuality in the fungus Penicillium roqueforti. Corradi N, editor. PLoS ONE. 2012;7:e49665.
Ropars J, Cruaud C, Lacoste S, Dupont J. A taxonomic and ecological overview of cheese fungi. International Journal of Food Microbiology. 2012;155:199–210.
Giraud F, Giraud T, Aguileta G, Fournier E, Samson R, Cruaud C, et al. Microsatellite loci to recognize species for the cheese starter and contaminating strains associated with cheese manufacturing. International Journal of Food Microbiology. 2010;137:204–13.
